Przykładowy przepływ pracy: Uruchamianie zadań EO Processing MPI na klastrze SLURM w chmurze NSIS
Celem tego artykułu jest zademonstrowanie uruchamiania zadań MPI na klastrze SLURM w chmurze.
MPI - Message Passing Interface - to standard komunikacji pomiędzy węzłami obliczeniowymi w architekturze obliczeń równoległych. Program napisany przy użyciu implementacji MPI umożliwia zlecanie każdemu węzłowi wykonania określonej części rozproszonego zadania. Węzły mogą następnie informować się nawzajem o stanie swoich konkretnych zadań, dzięki czemu na przykład jeden węzeł może kontynuować pracę w miejscu, w którym inny ją przerwał. Z punktu widzenia programisty, kod MPI jest napisany w centralnym skrypcie, tak jakby był napisany dla pojedynczej maszyny.
Co zostanie omówione?
Instalacja mpi4py – implementacji OpenMPI w Pythonie
Instalacja snappy – biblioteki Pythona do analizy i przetwarzania obrazów satelitarnych.
Instalacja dodatkowych modułów Pythona - s3cmd, boto3 i numpy.
Instalacja sieci danych EO we wszystkich węzłach, a tym samym dodanie do klastra możliwości obsługi danych EO.
Wdrożenie rozproszonego zadania MPI w klastrze
Uruchomienie tego zadania MPI w celu prostego wstępnego przetwarzania zdjęć satelitarnych
Pobranie i wyświetlenie jednego z obrazów przetworzonych w klastrze SLURM
Wymagania wstępne
Nr 1 Konto
Jest wymagane konto hostingowe NSIS z dostępem do interfejsu Horizon: https://horizon.cloudferro.com.
Nr 2 Uruchomiony klaster SLURM
Użyjemy konfiguracji klastra z artykułu:
Przykładowy klaster SLURM w chmurze NSIS z ElastiCluster.
Ten klaster ma jedem węzeł główny i 4 węzły robocze, nazywa się myslurmcluster i ma katalog /home udostępniony jako udział NFS na wszystkich węzłach. Ponadto na węzłach SLURM będzie zainstalowany system Ubuntu 18.04 z uruchomionym środowiskiem Python 3.6, które jest idealne dla SNAPPY.
Dostosuj poniższe polecenia i skrypty do docelowej instalacji SLURM.
Nr 3 Podstawowa znajomość ogólnego programowania
Pewne doświadczenie w korzystaniu z systemu Linux i programowaniu za pomocą języka takiego jak Python oraz korzystaniu z bibliotek lub oprogramowania takiego jak: OpenMPI, Snappy, mpi4py.
Więcej informacji można znaleźć pod tymi linkami:
Nr 4 Instalacja SNAP i s3cmd
Dodatkowo potrzebne będą moduły SNAP i s3cmd, które zostaną pobrane i zainstalowane w dalszej części artykułu.
Krok 1 Instalacja mpi4py
OpenMPI jest jednym ze standardów implementacji MPI. Jest on już wstępnie zanstalowany na klastrze SLURM, jeśli instalacja została przeprowadzona zgodnie z zaleceniami w sekcji Wymagania wstępne nr 2.
Uzyskajmy dostęp do węzła głównego i upewnijmy się, że znajdujemy się w katalogu /home/eouser:
elasticluster ssh myslurmcluster
cd /home/eouser
Należy pamiętać, że /home jest udziałem NFS na węźle głównym, który jest również zamontowany (z podkatalogami) na węzłach roboczych. Tak więc aktualizacja zawartości tego katalogu na węźle głównym sprawia, że jest ona również dostępna dla węzłów roboczych.
Uruchomimy następujące polecenia (z węzła głównego), aby zainstalować mpi4py, zarówno na węźle głównym, jak i na węzłach roboczych. Do instalacji na węzłach roboczych jest używane polecenie srun z flagą –nodes :
sudo apt install python3-mpi4py
srun --nodes=4 sudo apt install python3-mpi4py
Następnie utwórzmy minimalny program weryfikujący instalację mpi4py
touch mpi4py_hello.py
nano mpi4py_hello.py
mpi4py_hello.py
from mpi4py import MPI
comm = MPI.COMM_WORLD
print("Hello, my rank is: " + str(comm.rank))
Uruchom go za pomocą następującego polecenia:
mpirun -n 4 python3 mpi4py_hello.py
Rezultat będzie taki:
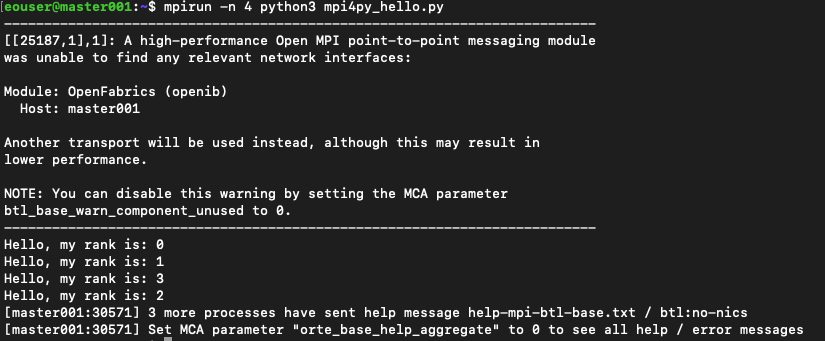
Jest wyświetlone ostrzeżenie o parametrze OpenFabrics i MCA. W przykładach w tym artykule zignorujemy takie ostrzeżenia. Istnieją również dwa sposoby na ich wyeliminowanie; jednym z nich jest użycie parametru
--mca btl_base_warn_component_unused 0
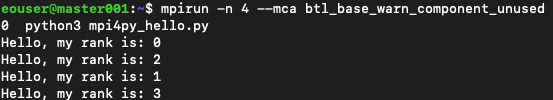
Polecenie byłoby wtedy takie:
mpirun -n 4 --mca btl_base_warn_component_unused 0 python3 mpi4py_hello.py
a wynik jest znacznie jaśniejszy:
Innym sposobem na trwałe wyłączenie ostrzeżenia jest edycja pliku /etc/openmpi/openmpi-mca-params.conf i wstawienie wiersza btl_base_warn_component_unused = 0.
Krok 2 Instalacja snappy
Snappy to biblioteka rozszerzeń Pythona dla aplikacji na komputer SNAP, udostępnionej przez ESA (Europejską Agencję Kosmiczną) do analizy i przetwarzania zdjęć satelitarnych.
Można pobrać najnowszą wersję ze strony pobierania SNAP i uruchomić pobieranie za pomocą wget przy użyciu poniższego polecenia. Następnie należy nadać temu plikowi atrybut „wykonywalny”:
wget https://download.esa.int/step/snap/9.0/installers/esa-snap_all_unix_9_0_0.sh
chmod +x esa-snap_all_unix_9_0_0.sh
Uruchomimy instalator w trybie nienadzorowanym. W ten sposób możemy zautomatyzować proces, unikając pytań interaktywnego instalatora o konfigurację każdego węzła.
W tym celu przygotujemy plik odpowiedzi (.varfile) i uruchomimy instalator z opcją -q. Plik można zmodyfikować zgodnie z własnymi preferencjami, na przykład w celu włączenia innych rozszerzeń.
Plik .varfile powinien znajdować się w tej samej lokalizacji co plik wykonywalny instalatora (patrz poniżej).
touch esa-snap_all_unix_9_0_0.varfile
nano esa-snap_all_unix_9_0_0.varfile
esa-snap_all_unix_9_0_0.varfile
executeLauncherWithPythonAction$Boolean=true
extendPathEnvVar$Boolean=true
forcePython$Boolean=true
pythonExecutable=/usr/bin/python3.6
sys.component.3109$Boolean=true
sys.component.RSTB$Boolean=true
sys.component.S1TBX$Boolean=true
sys.component.S2TBX$Boolean=true
sys.component.S3TBX$Boolean=true
Gdy plik odpowiedzi będzie gotowy, uruchom program instalacyjny za pomocą tego polecenia:
./esa-snap_all_unix_9_0_0.sh -q
Następnym krokiem jest włączenie snappy dla naszej dystrybucji Pythona. Uruchom z węzła głównego następujące polecenia:
cd ~/snap/bin
./snappy-conf /usr/bin/python3.6
Wynik jest następujący:
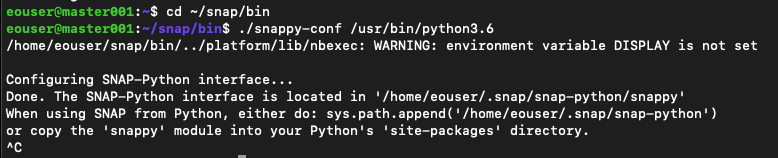
Po wyświetleniu komunikatu „Done” możesz przerwać operację za pomocą kombinacji klawiszy Ctrl+C.
Skopiujemy również moduł snappy do katalogu site-packages Pythona, aby umożliwić uruchamianie skryptów z dowolnej lokalizacji w drzewie katalogów (zarówno na węźle głównym, jak i węzłach roboczych).
sudo cp ~/.snap/snap-python/snappy /usr/lib/python3/dist-packages -r
srun --nodes=4 sudo cp ~/.snap/snap-python/snappy /usr/lib/python3/dist-packages -r
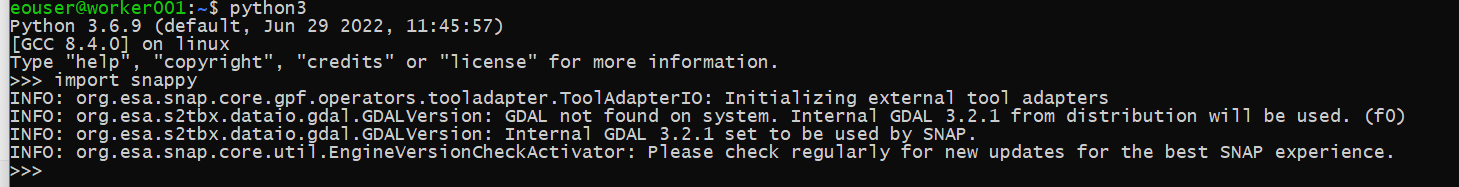
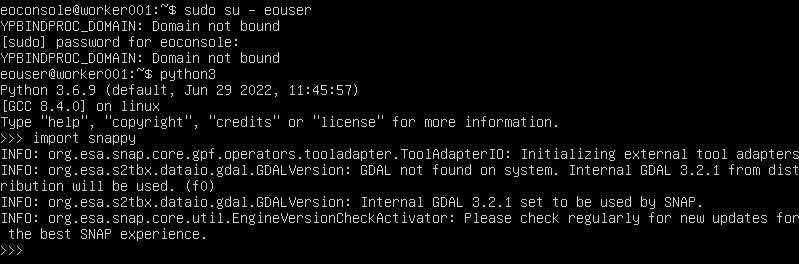
Będąc nadal na węźle głównym, sprawdź, czy to działa, w tym celu przejdź do konsoli Pythona i spróbuj zaimportować snappy:
python3
>>> import snappy
Jeśli wszystko pójdzie dobrze, powinniśmy zobaczyć ekran podobny do tego poniżej:
Powtórz tę czynność również na jednym z węzłów roboczych. Najprostszym sposobem jest użycie konsoli w Horizon; kliknij nazwę instancji węzła roboczego, a następnie zaloguj się w konsoli jako użytkownik eoconsole, wprowadź hasło i postępuj zgodnie z poleceniami, jak na poniższej ilustracji:
Krok 3 Dodanie możliwości obsługi danych EO do klastra
Dodaj sieć danych EO do węzłów głównych i roboczych.
Nasz skrypt snappy będzie pobierał zdjęcia satelitarne z repozytorium danych EO. Dlatego powinniśmy dodać sieć danych EO do naszych węzłów głównych i roboczych.
Teraz wyloguj się z maszyny klastra:
exit
Wpisz ze stacji roboczej następujące polecenia, pamiętając o zastąpieniu eodata_00341_3 nazwą twojej przypisanej sieci danych EO. Aby zobaczyć dokładną nazwę tej sieci, użyj poleceń menu Network -> Networks:
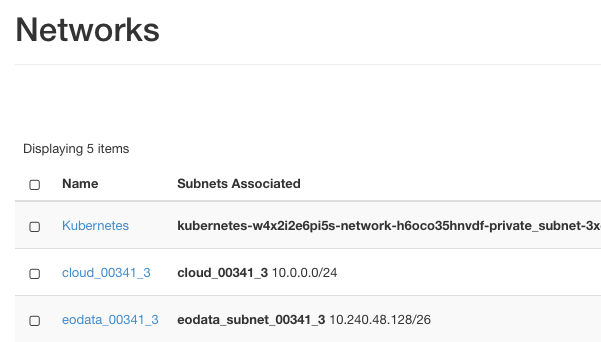
Z ilustracji wynika, że potrzebna nam nazwa to eodata_00341_3, a polecenia są następujące:
openstack server add network myslurmcluster-master001 eodata_00341_3
for i in `seq 1 4`; do openstack server add network myslurmcluster-worker00$i eodata_00341_3; done
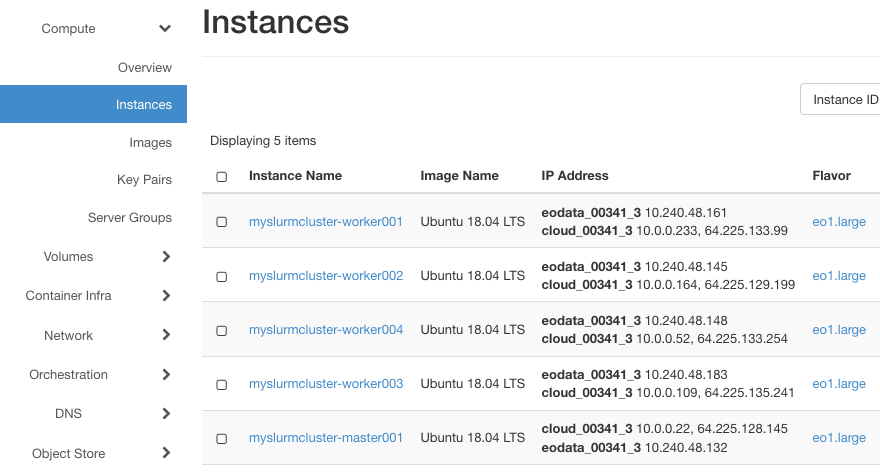
Jeśli operacja przebiegła prawidłowo, w Horizon zobaczysz, że dane EO zostały pomyślnie dodane do maszyn:
Następnie zaloguj się ponownie do węzła głównego za pomocą polecenia:
elasticluster ssh myslurmcluster
Instalacja pakietu s3cmd
Zainstaluj pakiet s3cmd do pobierania produktów SAFE z repozytorium danych EO:
sudo apt install -f s3cmd
srun --nodes=4 sudo apt install -f s3cmd
Musimy skonfigurować s3cmd do korzystania z klastra danych EO. W tym celu należy uruchomić polecenie:
s3cmd --configure
i postępuj zgodnie z instrukcjami kreatora, używając tych wartości:
klucz dostępu, klucz tajny: dowolny ciąg znaków
domyślny region: Region1
Punkt końcowy S3: eodata.nsiscloud.polsa.gov.pl
HTTPS: Nie
W przypadku innych wartości wystarczy wybrać wartości domyślne (nacisnąć Enter), a na koniec zapisać konfigurację.
Krok 4 Instalacja dodatkowych modułów Pythona
Na koniec zainstaluj dodatkowe moduły Pythona dla naszego skryptu przetwarzania danych: numpy i boto3. Po zainstalowaniu tych pakietów w przestrzeni użytkownika zostaną one również zainstalowane na węzłach roboczych za pośrednictwem udziału NFS. W tym celu z węzła głównego należy uruchomić następujące polecenia:
python3 -m pip install boto3
python3 -m pip install numpy
Krok 5 Uruchomienie zadania MPI przetwarzania obrazu
Nasz przykładowy skrypt pobiera obrazy satelitarne w formacie SAFE z repozytorium danych EO i wykonuje podstawowe operacje przetwarzania EO przy użyciu SNAP/Snappy. Przepływ pracy wykonuje następującą sekwencję:
Podzbiór produktów danych EO jest wyświetlany tylko w jednym z węzłów przy użyciu boto3.
Lista zostaje podzielona na fragmenty, a funkcja scatter MPI rozdziela fragmenty między węzły robocze
Każdy węzeł roboczy pobiera swój podzbiór produktów za pomocą s3cmd
Następnie każdy węzeł roboczy wykonuje przetwarzanie obrazu. Wybór narzędzi do przetwarzania służy jedynie do celów ilustracyjnych: używamy funkcji Resample SNAP, a następnie funkcji Subset.
Po przetworzeniu obrazu wynikowe pliki TIFF są zapisywane we wspólnym katalogu NFS.
Utwórz i wyedytuj plik image_processing.py:
touch image_processing.py
nano image_processing.py
Następnie wprowadź następującą treść:
image_processing.py
import boto3 import os import numpy as np from mpi4py import MPI import snappy from snappy import ProductIO from snappy import HashMap from snappy import GPF # MPI's retrieved parameters: number of all nodes on the cluster (size), and the current node (rank) comm = MPI.COMM_WORLD rank = comm.Get_rank() size = comm.Get_size() # boto3 setup: authorization and information about the subset of EO data repository to use (here Sentinel-2 L1C) s3_resource = boto3.resource('s3', aws_access_key_id='ANYKEY', aws_secret_access_key='ANYKEY', endpoint_url='http://eodata.nsiscloud.polsa.gov.pl') s3_client = s3_resource.meta.client bucket_name = 'DIAS' prefix = 'Sentinel-2/MSI/L1C/2022/10/01/' max_keys = 8 # using boto3 we generate a list of products, the list is generated only on one of the nodes (MPI's comm.rank==0) # we split the list into chunks, then the MPI's comm.scatter function distributes the chunks between all nodes sendbuf = [] if comm.rank == 0: collection_dicts = s3_client.list_objects(Delimiter='/', Bucket=bucket_name, Prefix=prefix, MaxKeys=max_keys)['CommonPrefixes'] collections = np.array([i['Prefix'] for i in collection_dicts]) chunked_collections = np.array_split(collections, size) sendbuf = chunked_collections collections_chunk = comm.scatter(sendbuf, root=0) # download .SAFE product files to local folder using s3cmd for col in collections_chunk: product_ex_prefix = col.replace(prefix, '') cmd = 'mkdir ' + product_ex_prefix os.system(cmd) cmd = 's3cmd get ' + '--recursive s3://EODATA/' + col + ' ~/' + product_ex_prefix os.system(cmd) # Read product to SNAP and apply SNAP's Resampling product = ProductIO.readProduct(product_ex_prefix) paramsRes = HashMap() paramsRes.put('targetResolution',20) productRes = GPF.createProduct('Resample', paramsRes, product) # Apply SNAP's Subset and save the file back to local folder paramsSub = HashMap() paramsSub.put('sourceBands', 'B2,B3,B4') paramsSub.put('copyMetadata', 'true') productSub = GPF.createProduct('Subset', paramsSub, productRes) ProductIO.writeProduct(productSub, product_ex_prefix, 'GeoTiff')
Uruchom skrypt za pomocą następującego polecenia:
mpirun --n 4 python3 image_processing.py
Po zakończeniu operacji możemy zobaczyć 8 pobranych produktów Sentinel-2 SAFE i 8 wygenerowanych plików tif.
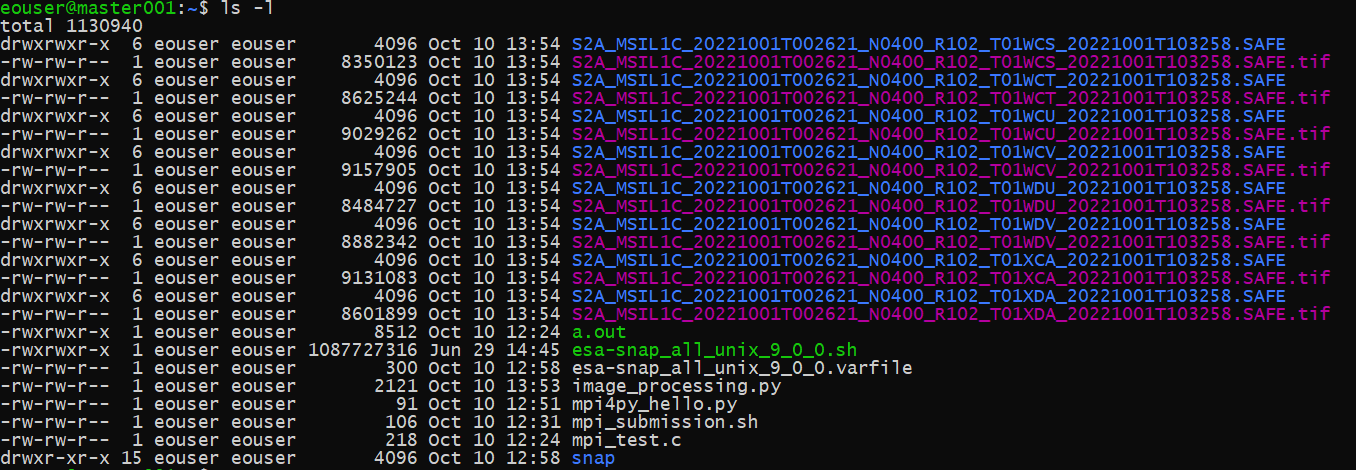
Co można zrobić dalej?
Jeśli używasz klastra SLURM do pracy z danymi satelitarnymi, możesz pobrać obrazy utworzone powyżej, używając polecenia takiego jak:
elasticluster sftp myslurmcluster
Aby uzyskać pełną perspektywę, należy również użyć dedykowanego oprogramowania, takiego jak SNAP desktop, aby wyświetlić obrazy w zakresie pasma widocznego dla ludzi.